如何选择核小体(nucleosomes)?
作者:Ellen N. Weinzapfel 博士,Lu Sun 博士

染色质及其调控在包括癌症等多种人类疾病中都发挥重要作用,并共同构成了学术、工业和临床研究中快速发展的研究领域1。染色质结构的基本亚单位是核小体,由大约147bp的DNA缠绕在核心组蛋白八聚体上构成(图1)2。组蛋白亚基有多种化学修饰,如甲基化、乙酰化和磷酸化,这些修饰被称为组蛋白修饰或翻译后修饰(PTMs)。
组蛋白修饰的功能:一种组蛋白编码
这些修饰调节染色质对各种蛋白质的可及性,如转录因子和核小体重塑复合物3,4。此外,组蛋白PTMs可以直接与染色质相互作用或招募染色质相互作用蛋白。因此,不同的组蛋白修饰与独特的基因组功能相关。例如,组蛋白H3赖氨酸27(H3K27me3)上的三甲基化与基因抑制密切相关5,而H3K4me3定位于活性转录起始位点,因此与基因激活相关6。
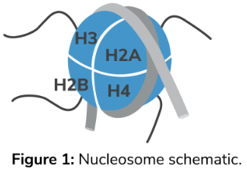
总之,这些组蛋白修饰网络及其动态调控协调了硬编码基因组的表观遗传控制,从而影响各种细胞过程,如基因调控、细胞分化和发育4,7。

研究组蛋白修饰的理想底物
越来越多的证据表明,核小体是体外表征许多染色质调节因子的最佳底物。这与之前的研究不同,之前的研究通常使用修饰的组蛋白肽来识别染色质抑制剂或定义新的组蛋白PTM相互作用。
在之前一篇关于组蛋白肽和核小体用于组蛋白PTM抗体验证的博客文章(https://www.epicypher.com/resources/blog/histone-peptides-vs-nucleosomes-which-is-better-for-chip-antibody-validation/)中,我们已经讨论了这个主题。然而,这些问题也与染色质修饰酶的研究有关。例如,NSD2甲基转移酶(与多发性骨髓瘤中的致癌重编程相关)需要核小体底物才能发挥体外活性8。同样,当使用重组核小体时,通过SETD8甲基转移酶的Km显示SAM辅因子比使用组蛋白肽底物时降低了10000倍9。
随着携带完全确定组蛋白修饰的重组核小体的出现,为下一代染色质研究提供了新的或更好的方法(如染色质结合蛋白分析、酶分析、抗体谱分析)。
如何获得重组核小体?
获得重组核小体的制造工艺有很多种,如EpiCypher通过设计来获得核小体(EpiCypher designer nucleosomes, dNucs)。一般来说,大部分相关的方法基本遵循相同的一系列步骤:

然而,并非所有的重组核小体都具有相同的质量!不同供应商的合成过程和质量验证标准可能存在很大差异。
如何挑选优质的核小体?
如果您要使用重组核小体进行染色质实验,则需考虑以下几点:
1、用于开发修饰组蛋白的方法。确保使用无疤痕方法整合所需的PTMs是很重要的,这种方法可以重现天然组蛋白结构。EpiCypher 使用几种不同的方法获得修饰的组蛋白,所有方法都会无疤痕整合组蛋白修饰。
为什么这很重要呢?许多市售的重组核小体是使用组蛋白PTM类似物构建的,如甲基赖氨酸类似物(MLAs) 10,其会导致修饰位点的氨基酸序列发生变化。这些非天然组蛋白修饰类似物已被证明会破坏与染色质调节蛋白和组蛋白PTM特异性抗体的相互作用,并且这对研究生理机制来讲是不理想的。因此,使用这些方法合成的核小体时应格外谨慎11-13。
2、修饰组蛋白的纯度。组装完全定义的同质核小体的下一步是对修饰组蛋白进行严格的质量控制。
在质控结果中,HPLC 迹线应显示是单一洗脱物质,表明组蛋白纯度>95%;平行高分辨率质谱(HRMS)应在预期质量的1道尔顿范围内显示一个单峰,没有任何意义的额外电荷质量(m/z)信号(例如图2A)。EpiCypher的所有修饰组蛋白均通过HPLC和HRMS分析进行验证。
为什么这很重要呢?不必要的物质(如甲硫氨酸氧化)可能引起结构改变,并影响静电相互作用或疏水相互作用,从而损害下游核小体组装的效率。
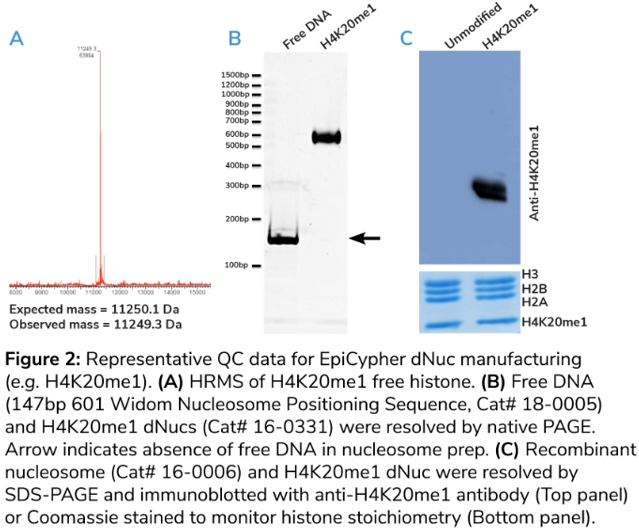
3、组装核小体的质量控制(QC)指标。DNA组装后核小体的质量验证对最终产品的信任保证至关重要。
EpiCypher使用天然PAGE分析DNA上的dNuc组装,其中使用约150bp DNA的高效组装应该只产生单一物质,这相对于未组装的游离DNA,其迁移率降低(图2B,下图)。
为什么这很重要呢?因为被污染的游离DNA会诱导染色质修饰酶(如NSD2)的异常活性,所以必须避免。此外,次优组装可能导致样品的异质性混合,包括错误定位的核小体。
我们还使用考马斯染色对最终的dNucs进行SDS-PAGE分析,以确保四种组蛋白的化学计量相等(图2C,下图)。然后,我们通过免疫印迹证实了整合组蛋白PTM的存在(图2C,上图)。
为什么这很重要呢?如果其他种类的蛋白质污染或偏离1:1:1:1的比例,则可能表明组蛋白降解或组装不良,因此有必要重新解析每个组蛋白。而免疫印迹对于确定修饰是否存在于组蛋白的正确位置上非常重要。
EpiCypher拥有令人印象深刻的产品目录,其中包括83个独特的重组核小体库存,并且有能力生产定制设计核小体,质量高、周期短,助力您的研究!
想了解更多关于EpiCypher重组核小体技术和产品的信息吗?请联系欣博盛!
EpiCypher核小体相关产品系列
◆ Recombinant Nucleosomes
◆ SNAP-ChIP® Spike-ins
◆ SNAP-CUTANA™ Spike-in Controls
◆ Modified Designer Nucleosomes (dNucs™)
◆ dCypher™ Nucleosome Panels
◆ EpiDyne® Chromatin Remodeling Substrates
◆ Recombinant Nucleosomes (rNucs)
◆ Mutant Nucleosomes
◆ Purified Nucleosomes (HeLa, Avian)
◆ Nucleosome Components and Subunits
◆ Variant Nucleosomes (vNucs)
◆ Methyl DNA Designer Nucleosomes
参考文献
1. Valencia AM, Kadoch C. Chromatin regulatory mechanisms and therapeutic opportunities in cancer. Nat Cell Biol, 2019. p. (PubMed PMID: 30602726)
2. Luger K, et al. Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature, 1997. 389(6648): p. 251-60. (PubMed PMID: 9305837)
3. Jenuwein T, Allis CD. Translating the histone code. Science, 2001. 293(5532): p. 1074-80. (PubMed PMID: 11498575)
4. Rothbart SB, Strahl BD. Interpreting the language of histone and DNA modifications. Biochim Biophys Acta, 2014. 1839(8): p. 627-43. (PubMed PMID: 24631868) (PMC4099259)
5. Cao R, et al. Role of histone H3 lysine 27 methylation in Polycomb-group silencing. Science, 2002. 298(5595): p. 1039-43. (PubMed PMID: 12351676)
6. Liang G, et al. Distinct localization of histone H3 acetylation and H3-K4 methylation to the transcription start sites in the human genome. Proc Natl Acad Sci U S A, 2004. 101(19): p. 7357-62. (PubMed PMID: 15123803) (PMC409923)
7. Bernstein BE, et al. A bivalent chromatin structure marks key developmental genes in embryonic stem cells. Cell, 2006. 125(2): p. 315-26. (PubMed PMID: 16630819)
8. Li Y, et al. The target of the NSD family of histone lysine methyltransferases depends on the nature of the substrate. J Biol Chem, 2009. 284(49): p. 34283-95.(PubMed PMID: 19808676)(PMC2797197)
9. Strelow JM, et al. The Use of Nucleosome Substrates Improves Binding of SAM Analogs to SETD8. J Biomol Screen, 2016. 21(8): p. 786-94.(PubMed PMID: 27369108)
10. Simon MD, et al. The site-specific installation of methyl-lysine analogs into recombinant histones. Cell, 2007. 128(5): p. 1003-12. (PubMed PMID: 17350582)(PMC2932701)
11. Gloss LM, Kirsch JF. Decreasing the basicity of the active site base, Lys-258, of Escherichia coli aspartate aminotransferase by replacement with gamma-thialysine. Biochemistry, 1995. 34(12): p. 3990-8. (PubMed PMID: 7696264)
12. Gellman SH. On the role of methionine residues in the sequence-independent recognition of nonpolar protein surfaces. Biochemistry, 1991. 30(27): p. 6633-6. (PubMed PMID: 2065050)
13. Seeliger D, et al. Quantitative assessment of protein interaction with methyl-lysine analogues by hybrid computational and experimental approaches. ACS Chem Biol, 2012. 7(1): p. 150-4. (PubMed PMID: 21991995) (PMC3265130)
EpiCypher的注册商标和知识产权可见链接:https://www.epicypher.com/intellectual-property/。
本文中的所有其他商标和商品均为其各自公司所有。
本文翻译自链接:https://www.epicypher.com/resources/blog/finding-the-best-substrate-for-studying-histone-modifications/,如与原文有出入的地方,请以英文原文为准。
未经EpiCypher公司事先书面同意,本文件不得部分或全部复制。
关于EpiCypher公司:
EpiCypher是一家成立于2012年的表观遗传学公司。从专有组蛋白肽阵列平台EpiGold™开始,EpiCypher开发了一系列同类产品。同时,EpiCypher是重组核小体制造和开发的全球领导者。利用其独有技术,不断增加产品库中高纯度修饰重组核小体(dNucs™)产品。dNuc™多样性的产品为破译组蛋白编码和加速药物开发提供了强大的工具。
EpiCypher还将dNuc™技术广泛的应用于多种分析测定产品中,包括:SNAP-ChIP®Spike-in Controls(用于抗体分析和ChIP定量), EpiDyne®底物(用于染色质重塑和抑制剂筛选及开发),dCyher™测定(用于探究表观遗传蛋白质-组蛋白PTM结合相互作用)。最近,EpiCypher还推出了针对ChIC、CUT&RUN和CUT&Tag的高灵敏度表观基因组图谱CUTANA™分析。

如需了解更多详细信息或相关产品,请联系EpiCypher中国授权代理商-欣博盛生物
全国服务热线: 4006-800-892 邮箱: market@neobioscience.com
深圳: 0755-26755892 北京: 010-88594029
上海: 021-34613729 广州: 020-87615159
代理品牌网站: www.neobioscience.com
自主品牌网站: www.neobioscience.net